1 litro di PBS: 8 gr NaCl; 0.2 gr KCl; 1.15 gr Na2HPO4 * 7H2O; 0.2 gr KH2PO4
Lavare la sospensione per 10 minuti in (0.2 M NaCl, 1 mM EDTA, 1 mM EGTA, and 10 mM HEPES (pH 7.5)).
Lavare le cellule tre volte con una soluzione fredda di PBS 1X, inibitore delle proteasi e inibitore delle fosfatasi. Aggiungere gli inibitori anche nei passi successivi.
Lisare le cellule con (50 mM Tris–HCl (pH 8.0), 1% (w/v) SDS, 10 mM EDTA).
Incubare a 0°C per 10 minuti.

Diluire la cromatina 1:7 nel buffer di diluizione (70 mM HEPES (pH 7.5), 2.5 mM NaCl, 1.5 mM EDTA, 1.5% Triton, and 0.6% deoxycholate).
Filtrare la sospensione in un filtro a siringa a maglie da 0.45 µm e raccogliere il filtrato in una provettina pre-raffreddata.

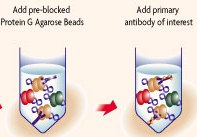
Incubare con 4–6 µg di anticorpo specifico a 4°C per l'intera notte.
A ogni provettina aggiungere 2 µl (0.33 µg/µl) di anticorpo AR(PG21).
Incubare per 12 ore a 4°C mantenendo in leggera agitazione.
Lavare le biglie due volte con buffer (50 mM HEPES (pH 7.5), 0.15 M NaCl, 1 mM EDTA, 1% Triton, 0.5% deoxycholate and 0.15% SDS).
Lavare con LNDET buffer (0.25 M LiCl, 1% NP40, 1% sodium deoxycholate, 1 mM EDTA, and 10 mM Tris (pH 8.0)).
Raccogliere le biglie mediante una veloce centrifugazione e lavarle 4 volte rispettivamente:
1) con il buffer freddo (20 mM Tris–HCl (pH 8.0), 150 mM NaCl, 2 mM Na-EDTA, 0.1% SDS and 1% TritonX-100). Recuperare le biglie mediante centrifugazione per 1 minuto a 24°C (4000g).
2) una volta a 24°C con il buffer (20 mM Tris–HCl (pH 8.0), 250 mM LiCl, 1 mM Na-EDTA, 0.5% IGEPAL (Sigma) non-ionic detergent and 0.5% sodium-deoxycholate (Sigma)). Recuperare le biglie mediante centrifugazione per 1 minuto a 24°C (4000g).
3) due volte con (10 mM Tris–HCl (pH 8.0), 1 mM Na-EDTA) a temperatura ambiente. Recuperare le biglie mediante centrifugazione per 1 minuto a 24°C (4000g).

Diluire con (10 mM Tris (pH 8.0), 1 mM EDTA).
Incubare per l'intera notte a 65°C per distaccare le proteine dal DNA.
Trattare con proteinasi K a 100 ng/µl e RNAase A per 1 ora a 55°C.
Incubare per 12 ore a 37°C.
Recuperare il supernatante mediante centrifugazione.
Incubare per 6 ore a 65°C per invertire il crosslinking.
Incubare per 12 ore a 37°C.
Recuperare il supernatante mediante centrifugazione.
Incubare per 6 ore a 65°C per invertire il crosslinking.
Estrarre in fenolo/cloroformio e precipitare in etanolo.
Risospendere il pellet in buffer TE.

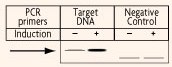
Gli amplificati sono stati estratti con fenolo, separati in gel di poliacrilamide al 6% e autoradiografati con lastra XAR-5.
Riferimenti:
Liqun Zhang, Mai Johnson, Kim H. Le, Makoto Sato, Romyla Ilagan, Meera Iyer, Sanjiv S. Gambhir, Lily Wu and Michael Carey
Interrogating Androgen Receptor Function in Recurrent Prostate Cancer.
Cancer Research 63, 4552-4560, August 1, 2003
Katsuaki Masudaa, Thomas Wernerb, Shilpi Maheshwaria, Matthias Frischb, Soyon Oha, Gyorgy Petrovicsa, Klaus Mayb, Vasantha Srikantana, Shiv Srivastavaa and Albert Dobia.
Androgen Receptor Binding Sites Identified by a GREF_GATA Model.
Journal of Molecular Biology. Volume 353, Issue 4, 4 November 2005, Pages 763-771
